머신러닝 문제는 훈련 샘플이 각각 수천에서 수백만 개의 특성을 가지고 있습니다.
이렇게 많은 특성은 훈련을 느리게 할 뿐 아니라, 좋은 솔루션을 찾기 어렵게 만듭니다.
이런 문제를 차원의 저주(Curse Of Dimensionality)라고 합니다.
차원을 축소 시키면 일부 정보가 유실됩니다. 그래서 훈련 속도가 빨라질 수는 있지만 시스템 성능이 조금 나빠질 수 있습니다. 또한 파이프라인이 조금 더 복잡하게 되고 유지 관리가 어려워집니다. 그러므로 차원 축소를 고려하기 전에 훈련이 너무 느린지 원본 데이터로 시스템을 훈련해봐야 합니다. 어떤 경우에는 훈련 데이터의 차원을 축소시키면 잡음이나 불필요한 세부사항을 걸러내므로 성능을 향상시킬 수 있습니다. 일반적으로는 훈련 속도만 빨라집니다.
훈련 속도를 높이는 것외에 차원 축소는 데이터 시각화(DataViz)에도 아주 유용합니다.
차원 수를 둘 또는 셋으로 줄이면 고차원 훈련 세트를 하나의 압축된 그래프로 그릴 수 있고 군집같은 시각적인 패턴을 감지해 중요한 통찰을 얻는 경우가 많습니다.
이번 챕터에서는 차원의 저주에 대해 논의하고 고차원 공간에서 어떤 일이 일어나느지 알아보겠습니다.
또한 차원 축소에 사용되는 두 가지 주요 접근 방법(투영과 매니폴드 학습), PCA, 커널 PCA, LLE를 다룹니다.
Chap8 목차
- 투영(Projection)
- 매니폴드 학습(Manifold Learning)
- 분산 보존
- 주성분
- d-차원으로 투영
- 적절한 차원 수 선택
- 압축을 위한 PCA
- 랜덤 PCA
- 점진적 PCA
- 커널 선택과 하이퍼파라미터 튜닝
- LLE
- 다른 기법
- 랜덤 투영
- MDS
- Isomap
- t-SNE
- LDA
1. 차원의 저주
○ 고차원은 많은 공간을 가지기 때문에(즉, 대부분의 훈련 데이터가 서로 멀리 떨어져 있기 때문에) 새로운 샘플도 훈련 샘플과 멀리 떨어져 있을 가능성이 높다
○ 이러한 경우 예측을 위해 훨씬 많은 외삽(Extrapolation)을 해야하기 때문에 저차원일 때보다 예측이 더 불안정
○ 즉, 훈련 세트의 차원이 클수록 과대적합 위험이 커짐
○ 이론적으로 차원의 저주를 해결하는 해결책
● 훈련 샘플의 밀도가 충분히 높아질 때까지 훈련 세트의 크기를 키우기
- 이러한 경우 일정 밀도에 도달하기 위해 필요한 훈련 샘플 수는 차원 수가 커짐에 따라 기하급수적으로 증가
2. 차원 축소를 위한 접근 방법
2-1 투영
○ 많은 특성은 거의 변화가 없는 반면, 다른 특성들은 서로 강하게 연관되어 있음
○ 결과적으로 모든 훈련 샘플이 고차원 공간 안의 저차원 부분 공간에 놓여있음
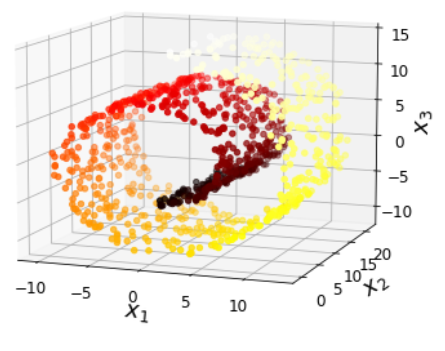
e.g. 원모양을 띤 3차원 데이터셋이 존재(왼쪽 그림)
● 모든 훈련 샘플이 거의 평면 형태로 놓여 있음
● 고차원(3D) 공간에 있는 저차원(2D) 부분 공간
● 여기서 모든 훈련 샘플을 이 부분 공간에 수직으로(샘플과 평면 사이의 가장 짧은 직선을 따라)
투영하면 오른쪽 그림과 같은 2D 데이터셋을 얻음
● 각 축은 (평면에 투영된 좌표인) 새로운 특성 z_1과 z_2에 대응


○ 차원 축소에 있어서 투영이 언제나 최선의 방법은 X. 많은 경우 아래 그림(왼쪽)에 표현된 스위스 롤 데이터셋처럼 부분 공간이 뒤틀리거나 휘어 있기도 함
○ 그냥 평면에 투영시키면(x_3를 버리면) 가운데 그림처럼 스위스 롤의 층이 서로 뭉개짐
● 우리가 원하는 것은 스위스 롤을 펼쳐서 오른쪽 그림처럼 2D 데이터셋을 얻는 것
[시각화 코드]
from sklearn.datasets import make_swiss_roll
X, t = make_swiss_roll(n_samples=1000, noise=0.2, random_state=42)- 왼쪽 사진
axes = [-11.5, 14, -2, 23, -12, 15]
fig = plt.figure(figsize=(6, 5))
ax = fig.add_subplot(111, projection='3d')
ax.scatter(X[:, 0], X[:, 1], X[:, 2], c=t, cmap=plt.cm.hot)
ax.view_init(10, -70)
ax.set_xlabel("$x_1$", fontsize=18)
ax.set_ylabel("$x_2$", fontsize=18)
ax.set_zlabel("$x_3$", fontsize=18)
ax.set_xlim(axes[0:2])
ax.set_ylim(axes[2:4])
ax.set_zlim(axes[4:6])
plt.show()
- 가운데와 오른쪽 사진
plt.figure(figsize=(11, 4))
plt.subplot(121)
plt.scatter(X[:, 0], X[:, 1], c=t, cmap=plt.cm.hot)
plt.axis(axes[:4])
plt.xlabel("$x_1$", fontsize=18)
plt.ylabel("$x_2$", fontsize=18, rotation=0)
plt.grid(True)
plt.subplot(122)
plt.scatter(t, X[:, 1], c=t, cmap=plt.cm.hot)
plt.axis([4, 15, axes[2], axes[3]])
plt.xlabel("$z_1$", fontsize=18)
plt.grid(True)
plt.show()
2-2 매니폴드 학습(Manifold Learning)
○ 스위스 롤은 2D 매니폴드의 한 예
● 2D 매니폴드는 고차원 공간에서 휘어지거나 뒤틀린 2D 모양
● d차원 매니폴드는 국부적으로 d차원 초평면으로 보일 수 있는 n차원 공간의 일부(d < n)
● 스위스 롤의 경우: d=2 & n=3 & 2차원으로 보이지만 3차원으로 말려있음
○ 많은 차원 축소 알고리즘이 훈련 샘플이 놓여 있는 매니폴드를 모델링하는 식으로 작동
● 근거: 실제 고차원 데이터셋이 더 낮은 저차원 매니폴드에 가깝게 놓여있다(매니폴드 가정| 매니폴드 가설)
○ 매니폴드 가정(Manifold Assumption)
● 병행되는 가정: 처리해야할 작업(회귀, 분류)이 저차원의 매니폴드 공간에 표현되면 더 간단해질 거라는 가정
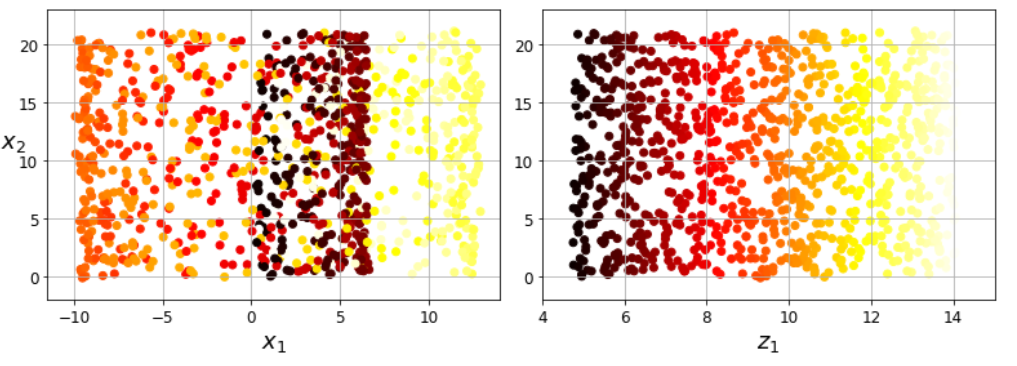
e.g. 아래 그림에서 첫 번째 행에서는 스위스 롤이 두 개의 클래스로 나뉘어 있음
- 3D(왼쪽)에서는 결정 경계가 매우 복잡하지만 펼쳐진 매니폴드 공간인 2D(오른쪽)에서는 결정 경계가 직선
● But 이런 암묵적인 가정이 항상 유효하지 않음
- 두 번째 행의 경우에는 결정 경계가 x_1 = 5에 있음(3D 공간에서는 매우 단순)
- 펼쳐진 매니폴드에서 결정 경계가 더 복잡해짐(네 개의 독립된 수직선)
$$ z_1=6, z_1=7, z_1=11.5, z_1=13.5 \textup{근처에 수직으로 나타남} $$
○ 요약
● 모델 훈련 전 훈련 세트의 차원을 감소시키면 훈련 속도는 빨라짐
● But 항상 더 낫거나 간단한 솔루션이 되는 건 아님(데이터셋에 달린 문제)




[시각화 코드]
from matplotlib import gridspec
axes = [-11.5, 14, -2, 23, -12, 15]
x2s = np.linspace(axes[2], axes[3], 10)
x3s = np.linspace(axes[4], axes[5], 10)
x2, x3 = np.meshgrid(x2s, x3s)
fig = plt.figure(figsize=(6, 5))
ax = plt.subplot(111, projection='3d')
positive_class = X[:, 0] > 5
X_pos = X[positive_class]
X_neg = X[~positive_class]
ax.view_init(10, -70)
ax.plot(X_neg[:, 0], X_neg[:, 1], X_neg[:, 2], "y^")
ax.plot_wireframe(5, x2, x3, alpha=0.5)
ax.plot(X_pos[:, 0], X_pos[:, 1], X_pos[:, 2], "gs")
ax.set_xlabel("$x_1$", fontsize=18)
ax.set_ylabel("$x_2$", fontsize=18)
ax.set_zlabel("$x_3$", fontsize=18)
ax.set_xlim(axes[0:2])
ax.set_ylim(axes[2:4])
ax.set_zlim(axes[4:6])
plt.show()
fig = plt.figure(figsize=(5, 4))
ax = plt.subplot(111)
plt.plot(t[positive_class], X[positive_class, 1], "gs")
plt.plot(t[~positive_class], X[~positive_class, 1], "y^")
plt.axis([4, 15, axes[2], axes[3]])
plt.xlabel("$z_1$", fontsize=18)
plt.ylabel("$x_2$", fontsize=18, rotation=0)
plt.grid(True)
plt.show()
fig = plt.figure(figsize=(6, 5))
ax = plt.subplot(111, projection='3d')
positive_class = 2 * (t[:] - 4) > X[:, 1]
X_pos = X[positive_class]
X_neg = X[~positive_class]
ax.view_init(10, -70)
ax.plot(X_neg[:, 0], X_neg[:, 1], X_neg[:, 2], "y^")
ax.plot(X_pos[:, 0], X_pos[:, 1], X_pos[:, 2], "gs")
ax.set_xlabel("$x_1$", fontsize=18)
ax.set_ylabel("$x_2$", fontsize=18)
ax.set_zlabel("$x_3$", fontsize=18)
ax.set_xlim(axes[0:2])
ax.set_ylim(axes[2:4])
ax.set_zlim(axes[4:6])
plt.show()
fig = plt.figure(figsize=(5, 4))
ax = plt.subplot(111)
plt.plot(t[positive_class], X[positive_class, 1], "gs")
plt.plot(t[~positive_class], X[~positive_class, 1], "y^")
plt.plot([4, 15], [0, 22], "b-", linewidth=2)
plt.axis([4, 15, axes[2], axes[3]])
plt.xlabel("$z_1$", fontsize=18)
plt.ylabel("$x_2$", fontsize=18, rotation=0)
plt.grid(True)
plt.show()3. PCA(Principal ComponentAnalysis; 주성분 분석)
○ 데이터에 가장 가까운 초평면(Hyperplane)을 정의 → 데이터를 이 평면에 투영
3-1 분산 보존
○ 저차원의 초평면에 훈련 세트를 투영하기 전에 올바른 초평면 선택이 우선
○ 아래의 그림의 경우
● 왼쪽 그래프: 간단한 2D 데이터셋이 세 개의 축(1차원 초평면)과 함께 표현됨
● 오른쪽 그래프: 데이터셋이 각 축에 투영된 결과
● 실선에 투영된 것: 분산을 최대로 보존
● 점선에 투영된 것: 분산을 매우 적게 유지
● 가운데 파선에 투영된 것: 분산을 중간 정도 유지
○ 다른 방향으로 투영하는 것보다 분산이 최대로 보존되는 축을 선택하는 것이 정보가 가장 적게 손실됨
● 원본 데이터셋과 투영된 것 사이의 평균 제곱 거리를 최소화하는 축

[시각화 코드]
angle = np.pi / 5
stretch = 5
m = 200
np.random.seed(3)
X = np.random.randn(m, 2) / 10
X = X.dot(np.array([[stretch, 0],[0, 1]])) # stretch
X = X.dot([[np.cos(angle), np.sin(angle)], [-np.sin(angle), np.cos(angle)]]) # rotate
u1 = np.array([np.cos(angle), np.sin(angle)])
u2 = np.array([np.cos(angle - 2 * np.pi/6), np.sin(angle - 2 * np.pi/6)])
u3 = np.array([np.cos(angle - np.pi/2), np.sin(angle - np.pi/2)])
X_proj1 = X.dot(u1.reshape(-1, 1))
X_proj2 = X.dot(u2.reshape(-1, 1))
X_proj3 = X.dot(u3.reshape(-1, 1))
plt.figure(figsize=(8,4))
plt.subplot2grid((3,2), (0, 0), rowspan=3)
plt.plot([-1.4, 1.4], [-1.4*u1[1]/u1[0], 1.4*u1[1]/u1[0]], "k-", linewidth=1)
plt.plot([-1.4, 1.4], [-1.4*u2[1]/u2[0], 1.4*u2[1]/u2[0]], "k--", linewidth=1)
plt.plot([-1.4, 1.4], [-1.4*u3[1]/u3[0], 1.4*u3[1]/u3[0]], "k:", linewidth=2)
plt.plot(X[:, 0], X[:, 1], "bo", alpha=0.5)
plt.axis([-1.4, 1.4, -1.4, 1.4])
plt.arrow(0, 0, u1[0], u1[1], head_width=0.1, linewidth=5, length_includes_head=True, head_length=0.1, fc='k', ec='k')
plt.arrow(0, 0, u3[0], u3[1], head_width=0.1, linewidth=5, length_includes_head=True, head_length=0.1, fc='k', ec='k')
plt.text(u1[0] + 0.1, u1[1] - 0.05, r"$\mathbf{c_1}$", fontsize=22)
plt.text(u3[0] + 0.1, u3[1], r"$\mathbf{c_2}$", fontsize=22)
plt.xlabel("$x_1$", fontsize=18)
plt.ylabel("$x_2$", fontsize=18, rotation=0)
plt.grid(True)
plt.subplot2grid((3,2), (0, 1))
plt.plot([-2, 2], [0, 0], "k-", linewidth=1)
plt.plot(X_proj1[:, 0], np.zeros(m), "bo", alpha=0.3)
plt.gca().get_yaxis().set_ticks([])
plt.gca().get_xaxis().set_ticklabels([])
plt.axis([-2, 2, -1, 1])
plt.grid(True)
plt.subplot2grid((3,2), (1, 1))
plt.plot([-2, 2], [0, 0], "k--", linewidth=1)
plt.plot(X_proj2[:, 0], np.zeros(m), "bo", alpha=0.3)
plt.gca().get_yaxis().set_ticks([])
plt.gca().get_xaxis().set_ticklabels([])
plt.axis([-2, 2, -1, 1])
plt.grid(True)
plt.subplot2grid((3,2), (2, 1))
plt.plot([-2, 2], [0, 0], "k:", linewidth=2)
plt.plot(X_proj3[:, 0], np.zeros(m), "bo", alpha=0.3)
plt.gca().get_yaxis().set_ticks([])
plt.axis([-2, 2, -1, 1])
plt.xlabel("$z_1$", fontsize=18)
plt.grid(True)
plt.show()3-2 주성분(PC)
○ 순서
● 훈련 세트에서 분산이 최대인 축을 찾음(바로 위 그림에서는 실선)
● 첫 번째 축에 직교하고 남은 분산을 최대한 보존하는 두 번째 축을 찾음(2D 예제서는 선택의 여지 없이 점선)
● (IF 고차원 데이터셋) PCA는 이전의 두축에 직교하는 세 번째 축을 찾으며 데이터셋에 있는 차원의 수만큼 축을 찾음
● i번째 축: 이 데이터의 i 번째 주성분(Principal Component; PC)
e.g. 위 그림에서
- 첫 번째 PC = 벡터 c_1이 놓인 축
- 두 번째 PC = 벡터 c_2가 놓인 축
○ 각 주성분을 위해 PCA는 주성분 방향을 가리키고 원점에 중앙이 맞춰진 단위 벡터를 찾음
○ 하나의 축에 단위 벡터가 반대 방향으로 2개 존재 So, PCA가 반환하는 단위 벡터의 방향은 일정하지 않음
(= 주성분의 방향은 일정치 않음)
○ 훈련 세트를 섞은 후 다시 PCA를 적용하면 새로운 PC 中 일부가 원래 PC와 반대 방향일 수 있음
(일반적으로 같은 축에 놓여 있을 것)
○ 어떤 경우에는 한 쌍의 PC가 회전하거나 서로 바뀔 수 있지만 보통은 같은 평면을 구성
○ 주성분 찾는 방법: 특잇값 분해(Singular Value Decomposition(SVD)라는 표준 행렬 분해 기술
● 훈련 세트 행렬 X를 세 개 행렬의 행렬 곱셈으로 분리 가능
[행렬 곱셈]
$$ U\sum V^{T} $$
● 찾고자 하는 모든 주성분의 단위 벡터가 V에 아래와 같이 담겨 있음
● (m,m) (m,n) (n,n), m=샘플개수, n=특성 개수
● V: 주성분
[주성분 행렬]
※ | = 열벡터
$$ V = \bigl(\begin{smallmatrix} | & | & & | \\ c_1 & c_2 & ... & c_n \\ | & | & & | \\ \end{smallmatrix}\bigr) $$
※ 공분산 행렬의 고유 벡터는 분산이 어느 방향으로 큰지를 나타냄
● 중앙에 맞춰진 훈련 세트의 공분산 행렬은 아래와 같음
공분산 행렬을 데이터의 특이값 분해로 표현한 것
○ 핵심 : 공분산 행렬이 SVD를 통해 특이값의 제곱과 직교행렬(V)에 의해 정의됨
○ 사용: 주성분 분석(PCA)와 같은 차원 축소 기법에서 사용
○ V: 데이터의 주성분 방향
○ ∑^{2}: 각 주성분의 중요도
[중앙에 맞춰진 훈련 세트의 공분산 행렬]
$$ Cov = \frac{1}{n-1}X^{T}X = \frac{1}{n-1}(U\sum V^{T})^{T}(U\sum V^{T})= \frac{1}{n-1}(V \sum U^{T})(U \sum V^{T})=\frac{1}{n-1}V\sum^{2}V^{T}=V\frac{\sum^{2}}{n-1}V^{T} $$
○ 공분산 행렬 = 각 변수 간 상관관계
○ 데이터 행렬 X의 공분산행렬에서
● X: 중앙에 맞춰서 n * p 데이터 행렬(n=데이터 포인트 개수, p = 특성 수)
● X^{T}: X의 전치행렬(n * p)
● 공분산 행렬의 크기: p * p(각 성분 = 변수 간의 공분산)
○ 특이값 분해(SVD)
● SVD를 사용해 행렬 X를 분해
$$ X = U\sum V^{T} $$
● U : n * n의 직교 행렬(데이터 포인트의 방향을 나타냄)
● ∑ : n * p 직사각형 대각 행렬(대각 성분 = X의 특이값)
● V^{T}: p * p 직교 행렬(변수의 방향)
● ∑^{T} ∑: 대각 행렬(각 대각 성분은 특이값의 제곱 = ∑^2)
[같은 값]
$$ X^{T}X = (U\sum V^{T})^T(U\sum V^{T}) $$
$$ (U\sum V^{T})^{T}= (V \sum U^{T}) $$
$$ (V \sum U^{T})(U \sum V^T) = V\sum^{2}V^{T} $$
∴ V가 공분산 행렬의 고유 벡터이고 주성분
○ 사이킷런의 PCA는 scipy.linalg.svd 함수를 사용하여 주성분 V를 구함
○ But, scipy.linalg.svd나 numpy.linalg.svd 함수에서 반환되는 U, s, V 값 중 V가 SVD 공식(아래의 공식)의 V^{T}
So, 아래 코드에서 svd 함수로부터 얻은 Vt를 다시 전치하여 첫 번째와 두 번째 열을 주성분으로 추출
○ numpy의 svd() 함수를 사용해 훈련 세트의 모든 주성분을 구한 후 처음 두 개의 PC를 정의하는 두 개의 단위 벡터 추출
X_centered = X - X.mean(axis=0)
U, s, Vt = np.linalg.svd(X_centered)
c1 = Vt.T[:, 0]
c2 = Vt.T[:, 1]○ PCA는 데이터셋의 평균이 0이라고 가정
○ 사이킷런의 PCA 파이썬 클래스는 이 작업을 대신 처리해줌
○ PCA를 직접 구현하거나 다른 라이브러리를 사용한다면 데이터를 원점에 맞추는 것이 중요
3-3 d차원으로 투영하기
○ 주성분을 모두 추출 → 처음 d개의 주성분으로 정의한 초평면에 투영 → 데이터셋의 차원을 d 차원으로 축소 가능
○ 해당 초평면은 분산을 가능한 한 최대로 보존하는 투영임을 보장
e.g. [훈련 세트를 d차원으로 투영하기]
● 3D 데이터셋은 데이터셋의 분산이 가장 큰 첫 두 개의 주성분으로 구성된 2D 평면에 투영
● 결과: 이 2D 투영 ≒ 원본 3D 데이터셋
● 초평면에 훈련 세트를 투영하고 [d차원으로 축소된 데이터셋]을 얻기 위해서는 [훈련세트를 d차원으로 투영하기]
와 같이 행렬 X와 V의 첫 d열로 구성된 행렬 W_d를 행렬 곱셈하면 됨
[훈련 세트를 d차원으로 투영하기]
$$ X_{\textup{d-proj}} = XW_d $$
[d차원으로 축소된 데이터셋]
$$ X^{\textup{d-proj}} $$
※ [훈련 세트를 d차원으로 투영하기]에서
● 샘플수 = 60개 → 행렬 X = (60, 3)
● W_d의 크기: (3,2) → So, 투영된 X_{d-proj} 행렬 크기 = (60,2)
● 주성분은 원본 데이터 공간에서 어떤 방향을 나타내므로 입력 행렬 X의 차원 3과 동일
[Python 코드]
○ 첫 두 개의 주성분으로 정의된 평면에 훈련 세트를 투영 → PCA변환 완료
W2 = Vt.T[:, :2]
X2D = X_centered.dot(W2)
X2D_using_svd = X2D
3-4 설명된 분산의 비율(Explained Variance Ratio)
○ 사이킷런의 PCA 모델은 SVD 분해 방법을 사용하여 구현
● 아래 코드: PCA 모델을 사용해 데이터셋의 차원을 2로 줄이는 코드(사이킷런은 자동으로 데이터를 중앙에 맞춤)
● PCA 변환기를 데이터셋에 학습 → components_ 속성에 W^d의 전치가 담겨있음
e.g. 첫 번째 주성분을 정의하는 단위 벡터: pca.components_.T[:, 0]
from sklearn.decomposition import PCA
pca = PCA(n_components=2)
X2D = pca.fit_transform(X)○ 설명된 분산의 비율은 explained_variance_ratio 변수에 저장(전체 분산에서 차지하는 비율)
○ 각 주성분의 축을 따라 있는 데이터셋의 분산 비율을 나타냄
3-5 적절한 차원 수 선택하기
○ 축소할 차원 수를 임의로 정하기보다는 충분한 분산(e.g. 95%)이 될 때까지 더해야 할 차원 수를 선택하는 것이 간단
○ 데이터 시각화를 위해 차원을 축소하는 경우 일반적으로 차원을 2 or 3개로 줄이는 것이 일반적
○ 코드
● 차원을 축소하지 않고 PCA를 계산한 뒤 훈련 세트의 분산을 95%로 유지하는데 필요한 최소한의 차원 수를 계산
※ n_components를 지정하지 않으면 특성 수와 샘플 수 중 작은 값으로 설정
※ np.cumsum 함수: 입력 배열의 원소를 차례대로 누적한 배열을 반환
○ 코드와 또 다른 방법
● 설명된 분산을 차원 수에 대한 함수로 그리기(cumsum을 그래프로 그리기)
● 설명된 분산의 빠른 성장이 멈추는 변곡점이 존재
● 여기서는 차원을 약 100으로 축소해도 설명된 분산을 크게 손해보지 않을 것

데이터 로드
from sklearn.datasets import fetch_openml
mnist = fetch_openml('mnist_784', version=1, as_frame=False)
mnist.target = mnist.target.astype(np.uint8)
from sklearn.model_selection import train_test_split
X = mnist["data"]
y = mnist["target"]
X_train, X_test, y_train, y_test = train_test_split(X, y)
차원 수 계산
pca = PCA()
pca.fit(X_train)
cumsum = np.cumsum(pca.explained_variance_ratio_)
d = np.argmax(cumsum >= 0.95) + 1
d # 154
cumsum을 그래프로 그리기
plt.figure(figsize=(6,4))
plt.plot(cumsum, linewidth=3)
plt.axis([0, 400, 0, 1])
plt.xlabel("Dimensions")
plt.ylabel("Explained Variance")
plt.plot([d, d], [0, 0.95], "k:")
plt.plot([0, d], [0.95, 0.95], "k:")
plt.plot(d, 0.95, "ko")
plt.annotate("Elbow", xy=(65, 0.85), xytext=(70, 0.7),
arrowprops=dict(arrowstyle="->"), fontsize=16)
plt.grid(True)
plt.show()3-6 압축을 위한 PCA
○ 차원 축소 → 훈련 세트의 크기 감소
e.g. MNIST 데이터셋에 분산의 95%를 유지하도록 PCA 적용
● 특성 수: 784개 → 150개 정도
● 분산은 유지 But 데이터셋 = 원본 크기의 20% 미만
○ PCA 투영의 변환을 반대로 적용하여 784개의 차원으로 되돌릴 수 있음
● 투영에서 일정량의 정보(유실된 5%의 분산)를 잃어버림 So, 원본 데이터셋을 얻을 수는 없음
But 원본 데이터와 매우 비슷할 것
○ 재구성 오차(Reconstruction Error)
● 원본 데이터와 재구성된 데이터(압축 후 원복한 것) 사이의 평균 제곱 거리
○ 코드
● 154차원으로 압축 + inverse_transform() 메서드를 사용해 784차원으로 복원
● 아래 사진은 원본 훈련 세트(왼쪽)과 샘플을 압축 후 복원한 결과(오른쪽)
- 이미지 품질은 조금 손실 But 숫자 모양은 거의 온전한 상태
○ 역변환 공식(원본의 차원 수로 되돌리는 PCA 역변환)
$$ X_{\textup{recovered}}= X_{\textup{d-proj}}W_{d}^{T} $$
주성분의 전치 행렬인 V^T(or W_{d}^{T})가 components_ 변수에 저장되어 있음
So, inverse_transform() 메서드에서 압축된 데이터셋에 components_ 를 점곱
→ 원본 데이터셋에서 구한 평균을 더함
○ X_train의 크기가 (52500, 784)라고 가정
● PCA 변환 후 X_reduced의 크기: (52500, 154)
● 주성분의 전치인 components_ 의 크기: (154, 784)
● X_reduced와 components_의 점곱 결과: (52500, 784)
∴ 원본 데이터셋의 차원으로 복원

pca = PCA(n_components=154)
X_reduced = pca.fit_transform(X_train)
X_recovered = pca.inverse_transform(X_reduced)- 원본 훈련 세트 + 압축 후 복원한 결과 시각화
def plot_digits(instances, images_per_row=5, **options):
size = 28
images_per_row = min(len(instances), images_per_row)
# n_rows = ceil(len(instances) / images_per_row) 와 동일합니다:
n_rows = (len(instances) - 1) // images_per_row + 1
# 필요하면 그리드 끝을 채우기 위해 빈 이미지를 추가합니다:
n_empty = n_rows * images_per_row - len(instances)
padded_instances = np.concatenate([instances, np.zeros((n_empty, size * size))], axis=0)
# 배열의 크기를 바꾸어 28×28 이미지를 담은 그리드로 구성합니다:
image_grid = padded_instances.reshape((n_rows, images_per_row, size, size))
# 축 0(이미지 그리드의 수직축)과 2(이미지의 수직축)를 합치고 축 1과 3(두 수평축)을 합칩니다.
# 먼저 transpose()를 사용해 결합하려는 축을 옆으로 이동한 다음 합칩니다:
big_image = image_grid.transpose(0, 2, 1, 3).reshape(n_rows * size,
images_per_row * size)
# 하나의 큰 이미지를 얻었으므로 출력하면 됩니다:
plt.imshow(big_image, cmap = mpl.cm.binary, **options)
plt.axis("off")plt.figure(figsize=(7, 4))
plt.subplot(121)
plot_digits(X_train[::2100])
plt.title("Original", fontsize=16)
plt.subplot(122)
plot_digits(X_recovered[::2100])
plt.title("Compressed", fontsize=16)
plt.show()3-7 랜덤 PCA
○ svd_solver 매개변수를 "randomized"로 지정
→ 사이킷런은 랜덤 PCA라 부르는 확률적 알고리즘을 사용해 처음 d개의 주성분에 대한 근삿값을 찾음
○ 알고리즘의 계산 복잡도(완전한 SVD방식과 다름)
● 완전한SVD 방식 계산 복잡도
$$ O(m*n^{2}) + O(n^{3}) $$
● 랜덤 PCA 계산 복잡도
$$ O(m * n^{2}) + O(d^{3}) $$
So, d가 n보다 많이 작으면 완전 SVD보다 훨씬 빠름
○ svd_solver의 기본값="auto"
● IF (m or n) > 500 && d > (m or n)의 80% → 자동으로 랜덤 PCA 알고리즘 사용
● Else: 완전한 SVD 방식 사용
● 완전한 SVD 방식을 강제하려면 svd_solver 매개변수를 "full"로 지정
3-8 점진적 PCA(Incremental PCA; IPCA)
○ PCA 구현의 문제: SVD 알고리즘을 실행하기 위해 전체 훈련 세트를 메모리에 올려야 한다는 점
○ 훈련 세트를 미니배치로 나눔 → IPCA 알고리즘에 한 번에 하나씩 주입
● 훈련 세트가 클 때 유용 & 온라인으로 PCA를 적용할 수 있음
○ [방법 1] 코드
● MNIST 데이터셋을 넘파이의 array_split() 함수를 사용해 100개의 미니배치로 나누기
● 사이킷런의 IncrementalPCA 파이썬 클래스에 주입 → MNIST 데이터셋의 차원을 154개(이전과 동일)로 줄임
● partial_fit() 메서드를 미니배치마다 호출(전체 훈련 세트를 사용하는 fit() 메서드 아님)
from sklearn.decomposition import IncrementalPCA
n_batches = 100
inc_pca = IncrementalPCA(n_components=154)
for X_batch in np.array_split(X_train, n_batches):
print(".", end="")
inc_pca.partial_fit(X_batch)
X_reduced = inc_pca.transform(X_train)
# 압축 확인
# X_recovered_inc_pca = inc_pca.inverse_transform(X_reduced)○ [방법 2] 코드 : 하드 디스크의 이진 파일에 저장된 큰 배열을 메모리에 있는 것 처럼 다루기
● 넘파이의 memmap 파이썬 클래스를 사용 → 필요할 때 데이터를 메모리에 적재
● IncrementalPCA: 특정 순간에 배열의 일부만 사용 So, 메모리 문제 해결 가능
● 이 방식의 경우 fit() 메서드 사용 가능
filename = "my_mnist.data"
m, n = X_train.shape
X_mm = np.memmap(filename, dtype='float32', mode='write', shape=(m, n))
X_mm[:] = X_train
del X_mm
X_mm = np.memmap(filename, dtype="float32", mode="readonly", shape=(m, n))
batch_size = m // n_batches
inc_pca = IncrementalPCA(n_components=154, batch_size=batch_size)
inc_pca.fit(X_mm)
4. 커널 PCA
○ 5장에서 샘플을 매우 높은 고차원 공간(특성 공간)으로 암묵적으로 매핑하여 SVM의 비선형 분류와 회귀를 가능하게 하는 커널 트릭에 대해 설명
○ 고차원 특성 공간에서의 선형 결정 경계는 원본 공간에서는 복잡한 비선형 결정 경계에 해당
○ 같은 기법을 PCA에 적용해 차원 축소를 위한 복잡한 비선형 투형을 수행 가능(kPCA; kernel PCA)
○ 코드
● KernelPCA를 사용해 RBF 커널로 kPCA를 적용
from sklearn.decomposition import KernelPCA
rbf_pca = KernelPCA(n_components=2, kernel="rbf", gamma=0.04)
X_reduced = rbf_pca.fit_transform(X)○ 아래 그림은 (단순히 PCA를 사용한 것과 동일한) 선형/RBF/시그모이드 커널을 사용해 2차원으로 축소시킨 스위스 롤

[시각화 코드]
from sklearn.decomposition import KernelPCA
lin_pca = KernelPCA(n_components=2, kernel="linear", fit_inverse_transform=True)
rbf_pca = KernelPCA(n_components=2, kernel="rbf", gamma=0.0433, fit_inverse_transform=True)
sig_pca = KernelPCA(n_components=2, kernel="sigmoid", gamma=0.001, coef0=1, fit_inverse_transform=True)
y = t > 6.9
plt.figure(figsize=(11, 4))
for subplot, pca, title in ((131, lin_pca, "Linear kernel"), (132, rbf_pca, "RBF kernel, $\gamma=0.04$"), (133, sig_pca, "Sigmoid kernel, $\gamma=10^{-3}, r=1$")):
X_reduced = pca.fit_transform(X)
if subplot == 132:
X_reduced_rbf = X_reduced
plt.subplot(subplot)
#plt.plot(X_reduced[y, 0], X_reduced[y, 1], "gs")
#plt.plot(X_reduced[~y, 0], X_reduced[~y, 1], "y^")
plt.title(title, fontsize=14)
plt.scatter(X_reduced[:, 0], X_reduced[:, 1], c=t, cmap=plt.cm.hot)
plt.xlabel("$z_1$", fontsize=18)
if subplot == 131:
plt.ylabel("$z_2$", fontsize=18, rotation=0)
plt.grid(True)
plt.show()4-1 커널 선택과 하이퍼파라미터 튜닝
○ kPCA는 비지도 학습 So, 좋은 커널과 하이퍼파라미터를 선택하기 위한 명확한 성능 측정 기준 X
○ [방법 1] 코드
● 두 단계의 파이프라인 생성
- kPCA를 사용해 차원을 2차원으로 축소 & 분류를 위해 로지스틱 회귀 적용
- 파이프라인 마지막 단계에서 가장 높은 분류 정확도를 얻기 위해 GridSearchCV를 사용
∴ kPCA의 가장 좋은 커널과 gamma 파라미터를 찾음(best_params_ 변수에 저장됨)
from sklearn.model_selection import GridSearchCV
from sklearn.linear_model import LogisticRegression
from sklearn.pipeline import Pipeline
clf = Pipeline([
("kpca", KernelPCA(n_components=2)),
("log_reg", LogisticRegression(solver="lbfgs"))
])
param_grid = [{
"kpca__gamma": np.linspace(0.03, 0.05, 10),
"kpca__kernel": ["rbf", "sigmoid"]
}]
grid_search = GridSearchCV(clf, param_grid, cv=3)
grid_search.fit(X, y)
- kPCA의 가장 좋은 커널과 gamma 파라미터 찾기
print(grid_search.best_params_)
# {'kpca__gamma': 0.043333333333333335, 'kpca__kernel': 'rbf'}○ [방법 2] 코드(완전한 비지도 학습 방법)
● 가장 낮은 재구성 오차를 만드는 커널과 하이퍼파라미터 선택하는 방식
● 재구성은 선형 PCA만큼 쉽지 않음
● 아래 그림
- 왼쪽 위: 스위스 롤의 원본 3D 데이터셋
- 오른쪽 위: RBF 커널의 kPCA를 적용한 2D 데이터셋
- 커널 트릭 덕분에 이 변환은 특성 맵을 사용하여 훈련 세트를 무한 차원의 특성 공간(오른쪽 아래)로 매핑
→ 변환된 데이터셋을 선형 PCA를 사용해 2D로 투영한 것과 수학적으로 동일

○ 축소된 공간에 있는 샘플에 대해 선형 PCA를 역전시키면 재구성된 데이터 포인트는 원본 공간이 아닌 특성 공간에 놓임
● 특성공간 = 무한 차원 So, 재구성된 포인트 & 재구성에 따른 실제 에러를 계산 불가
● 재구성된 포인트에 가깝게 매핑된 원본 공간의 포인트를 찾을 수는 있음(재구성 원상; pre-image)
● 원상을 얻게 되면 원본 샘플과의 제곱 거리 측정이 가능
So, 재구성 원상의 오차를 최소화하는 커널과 하이퍼파라미터를 선택 가능
$$ 오차 = \sum_{i=1}^{n}(x_i - \hat{x}_i)^2 $$
$$ x_i = \textup{원본 데이터의 i 번째 샘플} $$
$$ \hat{x}_{i} = \textup{재구성된 데이터의 i번째 샘플} $$
$$ \textup{이 값이 작을수록, 원본 데이터와 재구성된 데이터의 차이가 적다는 뜻} $$
※ 원상(역상; Inverse Image): 어떤 함수(f: X → Y)에 대한 공역(Y) 원소에 대응하는 정의역(X) 원소로 이뤄진 부분집합
○ 재구성하는 방법
● 투영된 샘플을 훈련 세트로, 원본 세트를 타깃으로 하는 지도 학습 회귀 모델을 훈련시키기
● fit_inverse_transform=True로 지정하면 자동 수행
※ KernelPCA는 fit_inverse_transform=False가 기본값이면 inverse_transform() 메서드가 없음
fit_inverse_transform=True를 했을 때만 inverse_transform() 메서드 생성
rbf_pca = KernelPCA(n_components=2, kernel="rbf", gamma=0.0433,
fit_inverse_transform=True)
X_reduced = rbf_pca.fit_transform(X)
X_preimage = rbf_pca.inverse_transform(X_reduced)○ 재구성 원상 오차 계산
from sklearn.metrics import mean_squared_error
mean_squared_error(X, X_preimage)
# 32.786308795766104
5. LLE(Locally Linear Embedding; 지역적 선형 임베딩)와 다른 차원 축소 기법
○ LLE = 또 다른 강력한 비선형 차원 축소(NLDR;Nonlinear Dimensionality Reduction) 기술
○ 투영에 의존하지 않는 매니폴드 학습
○ 순서
● 각 훈련 샘플이 가장 가까운 이웃(c.n.; Closest Neighbor)에 얼마나 선형적으로 연관된지 측정
● 국부적인 관계가 가장 잘 보존되는 훈련 세트의 저차원 표현을 찾음
○ 장점: 특히 잡음이 너무 많지 않은 경우 꼬인 매니폴드를 펼치는 데 잘 작동
○ 결과 2D 데이터셋은 아래와 같다.
● 스위스 롤이 완전히 펼쳐짐
● 지역적으로는 샘플 간 거리가 잘 보존
● 크게 보면 샘플 간 거리가 잘 유지되지 않음
● 오른쪽은 압축, 왼쪽은 확장 But LLE는 매니폴드를 모델링하는 데 잘 동작

○ LLE 작동 방식
● 알고리즘이 각 훈련 샘플 x^{(i)}에 대해 가장 가까운 k개의 샘플을 찾음(코드에서는 k= 10)
● 이 이웃에 대한 선형 함수로 x^{(i)}를 재구성
● 구체적인 내용
$$ x^{(i)} \textup{와} \sum_{j=1}^{m} w_{i, j}x^{(j)} \textup{사이의 제곱 거리가 최소가 되는} w_{i,j} \textup{를 찾는 것} $$
$$ \textup{여기서} x^{(j)} \textup{가} x^{(i)} \textup{의 가장 가까운 k 개 이웃 중 하나가 아닐 경우} w_{i, j} = 0 \textup{이 됨} $$
● W = w_{i, j}를 모두 담은 가중치 행렬
● 두 번째 제약 = 각 훈련 샘플 x^{(i)}에 대한 가중치를 단순히 정규화 하는 것
[LLE 단계 1: 선형적인 지역 관계 모델링]
$$ \hat{W}=\underset{w}{\textup{argmin}}\sum_{i=1}^{m}(x^{(i)}-\sum_{j=1}^{m}w_{i,j}x^{(j)})^2 $$
$$ \textup{[condition]}\begin{cases}w_{i,j} & x^{(j)}\textup{가} x^{(i)}\textup{의 최근접 이웃 k개 중 하나가 아닐 때}\\
\sum_{j=1}^{m}w_{i,j}=1 & i = 1,2,...,m\textup{일때}\end{cases} $$
○ 이 단계를 거치면(가중치 \hat{w}_{i,j}를 담은) 가중치 행렬 \hat{W}은 훈련 샘플 사이에 있는 지역 선형 관계를 담음
○ 두 번째 단계
● 관계가 보존되도록 훈련 샘플을 d차원 공간(d < n)으로 매핑
$$ \textup{만약} z^{(i)}\textup{가 d차원 공간에서} x^{(i)} \textup{의 image라면 가능한} z^{(i)}\textup{와} \sum_{j=1}^{m}\hat{w}_{i,j}z^{(i)}\textup{사이의 거리가 최소화 되어야 함} $$
● 해당 아이디어는 LLE 단계 2 식과 같은 제약이 없는 최적화 문제로 바꾸어 줌
- 첫 번째 단계와 비슷해 보이지만 차이가 있음
● 샘플을 고정하고 최적의 가중치를 찾는 대신
● 반대로 가중치를 고정하고 저차원의 공간에서 샘플 이미지의 최적 위치를 찾음
$$ Z \textup{는 모든} z^{(i)} \textup{를 포함하는 행렬} $$
[LLE 단계 2: 관계를 보존하는 차원 축소]
$$ Z = \underset{z}{\textup{argmin}}\sum_{i=1}^{m}(z^{(i)}-\sum_{j=1}^{m}\hat{w}_{i,j}z^{(j)})^2 $$
○ 사이킷런이 제공하는 LLE 구현의 계산 복잡도
$$ \textup{k개의 가장 가까운 이웃을 찾기:} O(mlog(m)nlog(k)) $$
$$ \textup{가중치 최적화에} O(mnk^{3}) $$
$$ \textup{저차원 표면을 만드는데} O(dm^{2}) $$
마지막 항의 m^2 때문에 이 알고리즘을 대량의 데이터셋에 적용하기 어려움
[시각화 코드]
from sklearn.manifold import LocallyLinearEmbedding
lle = LocallyLinearEmbedding(n_components=2, n_neighbors=10, random_state=42)
X_reduced = lle.fit_transform(X)plt.title("Unrolled swiss roll using LLE", fontsize=14)
plt.scatter(X_reduced[:, 0], X_reduced[:, 1], c=t, cmap=plt.cm.hot)
plt.xlabel("$z_1$", fontsize=18)
plt.ylabel("$z_2$", fontsize=18)
plt.axis([-0.065, 0.055, -0.1, 0.12])
plt.grid(True)
save_fig("lle_unrolling_plot")
plt.show()5-2 다른 차원 축소 기법
5-2-1 랜덤 투영(Random Projection)
○ 랜덤한 선형 투영을 사용해 데이터를 저차원 공간으로 투영
○ 이러한 랜덤 투영이 실제로 거리를 잘 보존하는 것으로 밝혀짐
○ 차원 축소 품질 = 샘플 수와 목표 차원 수에 따라 다름
But 초기 차원수에는 의존적이지 않음
5-2-2 다차원 스케일링(MDS;Multidimensional Scaling)
○ 샘플 간의 거리를 보존하면서 차원을 축소
5-2-3 Isomap
○ 각 샘플을 가장 가까운 이웃과 연결하는 식으로 그래프 생성
○ 샘플 간의 지오데식 거리(Geodesic distance)를 유지하면서 차원 축소
※ 두 노드 사이의 지오데식 거리 = 두 노드 사이의 최단 경로를 이루는 노드의 수
5-2-4 t-SNE(t-distributed stochastic neighbor embedding)
○ 비슷한 샘플은 가까이, 비슷하지 않은 샘플은 멀리 떨어지도록 차원을 축소
○ 시각화에 주로 사용(특히, 고차원 공간에 있는 샘플의 군집을 시각화할 때 사용)
5-2-5 선형 판별 분석(LDA; Linear Discriminant Analysis)
○ 사실은 분류 알고리즘 But 훈련 과정에서 클래스 사이를 가장 잘 구분하는 축을 학습
○ 이 축은 데이터가 투영되는 초평면을 정의하는 데 사용 가능
○ 장점 : 투영을 통해 가능한 한 클래스를 멀리 떨어지게 유지
So, SVM 분류기 같은 다른 분류 알고리즘을 적용 전에 차원 축소 하기 좋음

[시각화 코드]
titles = ["MDS", "Isomap", "t-SNE"]
plt.figure(figsize=(11,4))
for subplot, title, X_reduced in zip((131, 132, 133), titles,
(X_reduced_mds, X_reduced_isomap, X_reduced_tsne)):
plt.subplot(subplot)
plt.title(title, fontsize=14)
plt.scatter(X_reduced[:, 0], X_reduced[:, 1], c=t, cmap=plt.cm.hot)
plt.xlabel("$z_1$", fontsize=18)
if subplot == 131:
plt.ylabel("$z_2$", fontsize=18, rotation=0)
plt.grid(True)
plt.show()'Networks > Hands-On 머신러닝 정리' 카테고리의 다른 글
| Hands-On Machine Learning 정리 - 머신러닝(Chapter 9: 비지도 학습) - Part. 2 (4) | 2024.10.22 |
|---|---|
| Hands-On Machine Learning 정리 - 머신러닝(Chapter 9: 비지도 학습) - Part. 1 (0) | 2024.10.22 |
| Hands-On Machine Learning 정리 - 머신러닝(Chapter 7: 앙상블 학습과 랜덤 포레스트) (0) | 2024.10.21 |
| Hands-On Machine Learning 정리 - 머신러닝(Chapter 6: 결정 트리) (0) | 2024.10.21 |
| Hands-On Machine Learning 정리 - 머신러닝(Chapter 5: SVM) (4) | 2024.10.20 |